Introductie
MNP is het Model for Nature Policy, voorheen de Meta Natuurplanner. MNP is bedoeld om te bepalen of bepaalde natuurlijke soorten (dwz geen gecultiveerde of gedomesticeerde) duurzaam in Nederland kunnen bestaan, gegeven bepaalde inrichting en kwaliteit van de leefomgeving. MNP is momenteel toegerust voor het doorrekenen van vogels, vlinders en planten. Het percentage soorten dat duurzaam kan bestaan wordt aangeduid met het doelbereik, dit is een van de belangrijkste MNP uitkomsten. Uit de Evaluatie Natuurpact volgde een doebereik van 53% (77 van 146 soorten, 16 vogels, 9 vlinders, 52 planten).
MNP is ontwikkeld door Wageningen Environmental Research (WENR) in opdracht van het Planbureau voor de Leefomgeving (PBL). Een belangrijke toepassingsveld van MNP zijn scenariostudies, waarbij bijvoorbeeld het areaal en kwaliteit natuurgebieden verandert; welke soorten profiteren daarvan en welke niet? Een voorbeeld hiervan is de Scenario Natuurinclusief studie. Tevens ligt MNP ten grondslag aan diverse indicatoren van het Compendium voor de Leefomgeving (CLO), zoals de Geschiktheid ruimtelijke condities landnatuur.
Eerdere versies van MNP zijn ontwikkeld in Access en C++. Diverse rapportages beschrijven MNP in detail:
Van Hinsberg et al. (2023) Review van de MetaNatuurplanner (MNP)
Pouwels et al. (2017) MetaNatuurplanner v4.0 - Status A: Toepassing voor Evaluatie Natuurpact
Pouwels et al. (2016) MetaNatuurplanner v2.0 Status A
Zie ook het PBL: MetaNatuurPlanner
In 2022 is besloten om MNP te herschrijven naar Python. De aanleiding hiertoe was:
toekomstbestendig maken van de broncode en overdragen naar volgende generatie onderzoekers
MNP gebruiksvriendelijker maken
MNP output laagdrempeliger en intuatiever maken
MNP voorbereiden op toekomstige uitbreiding van toepassingsgebied en rijkweidte
Eind 2022 is de herprogrammering van MNP afgerond, niettegenstaande diverse uitbreidingsideeen voor later. Uit [testen](#vergelijking-mnp-7.0-MNP 4.0) blijkt dat de nieuwe MNP uitkomsten naargenoeg identieke resultaten produceert als MNP 4.0, gegeven dezelfde input.
MNP in theorie
Het MNP berekent het verwachte effect van beleidsmaatregelen op het duurzaam voorkomen van soorten in Nederland. Het gaat hierbij om de grote lijnen, het model is een sterke versimpeling van de werkelijkheid en rekent voornamelijk met factoren waar beleid invloed op kan hebben.
Het gebruik van MNP voor scenario’s gebeurt in een breder framework (Pouwels et al., 2020), zoals hieronder uitgebeeld.
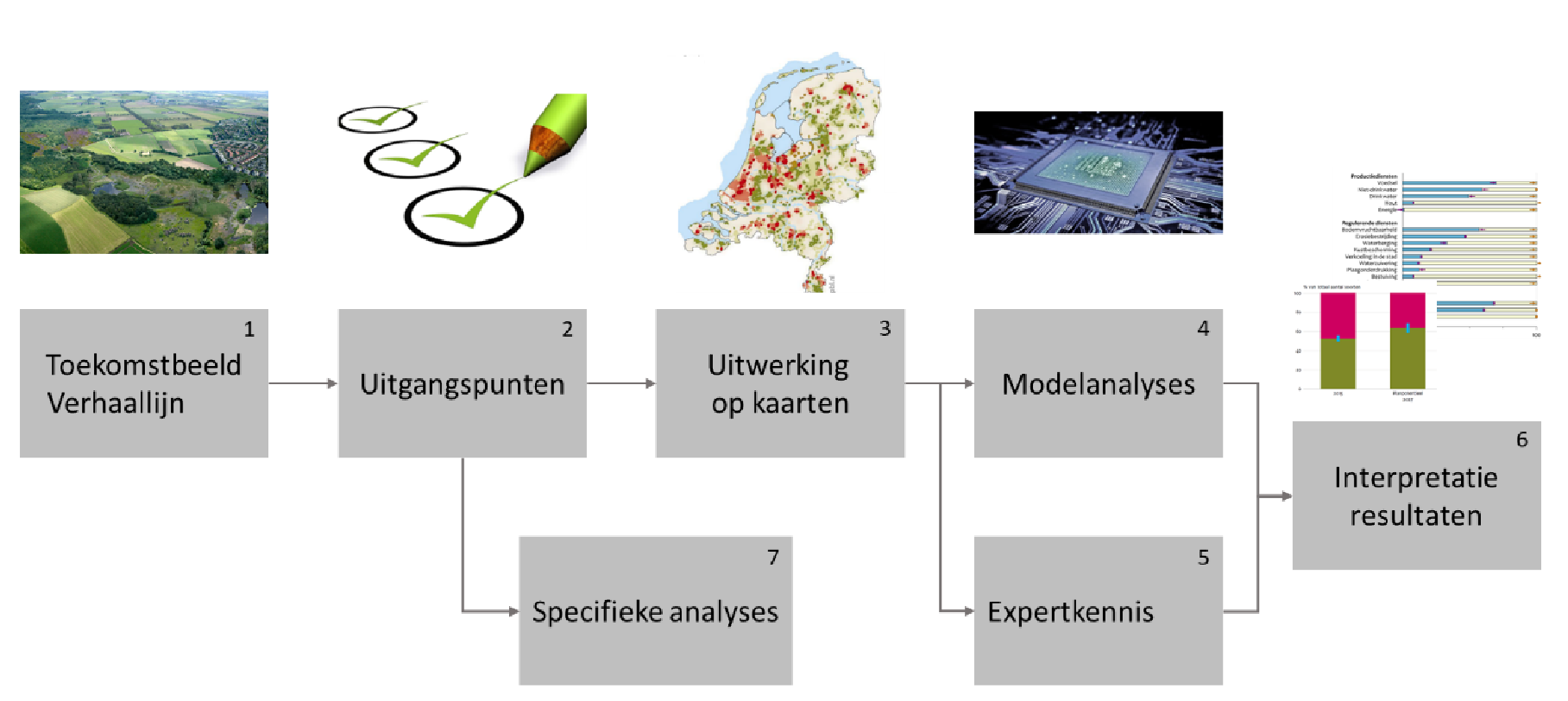
Het (rekenmodel) MNP zelf, beslaat hierin enkel fase 4, die weer is onderverdeeld in drie stappen:
Toekomstbeeld en verhaallijn
Uitgangspunten
Uitwerking op kaarten
Modelanalyses MNP
Determineren van soortspecifieke habitatgeschiktheid
Evaluatie van verwacht voorkomen per soort
Aggregeren naar beleidsrelevante indicatoren
Expertkennis
Interpretatie resultaten
Specifieke analyses
Van scenario naar invoerkaarten
Fases 1 t/m 3 van het framework vallen buiten het MNP en zijn grotendeels maatwerk. Breman et al. (2022) en Pouwels et al. (2020) beschrijven hoe deze fases voor de Natuurverkenning 2050 zijn doorlopen. Biersteker en Roelofsen (2024) beschrijven dezelfde fases voor een ex-ante evaluatie. Hoe deze fases precies worden uitgevoerd hangt dus af van het type analyse.
Fase 3, het uitwerken van uitgangspunten op kaarten volgt wel een redelijk vast patroon. Zie Scenariobuilder voor een uitleg over hoe deze fase wordt gefaciliteerd.
Modelanalyses MNP
Hieronder volgt een flowchart van de stappen in MNP (fase 4).
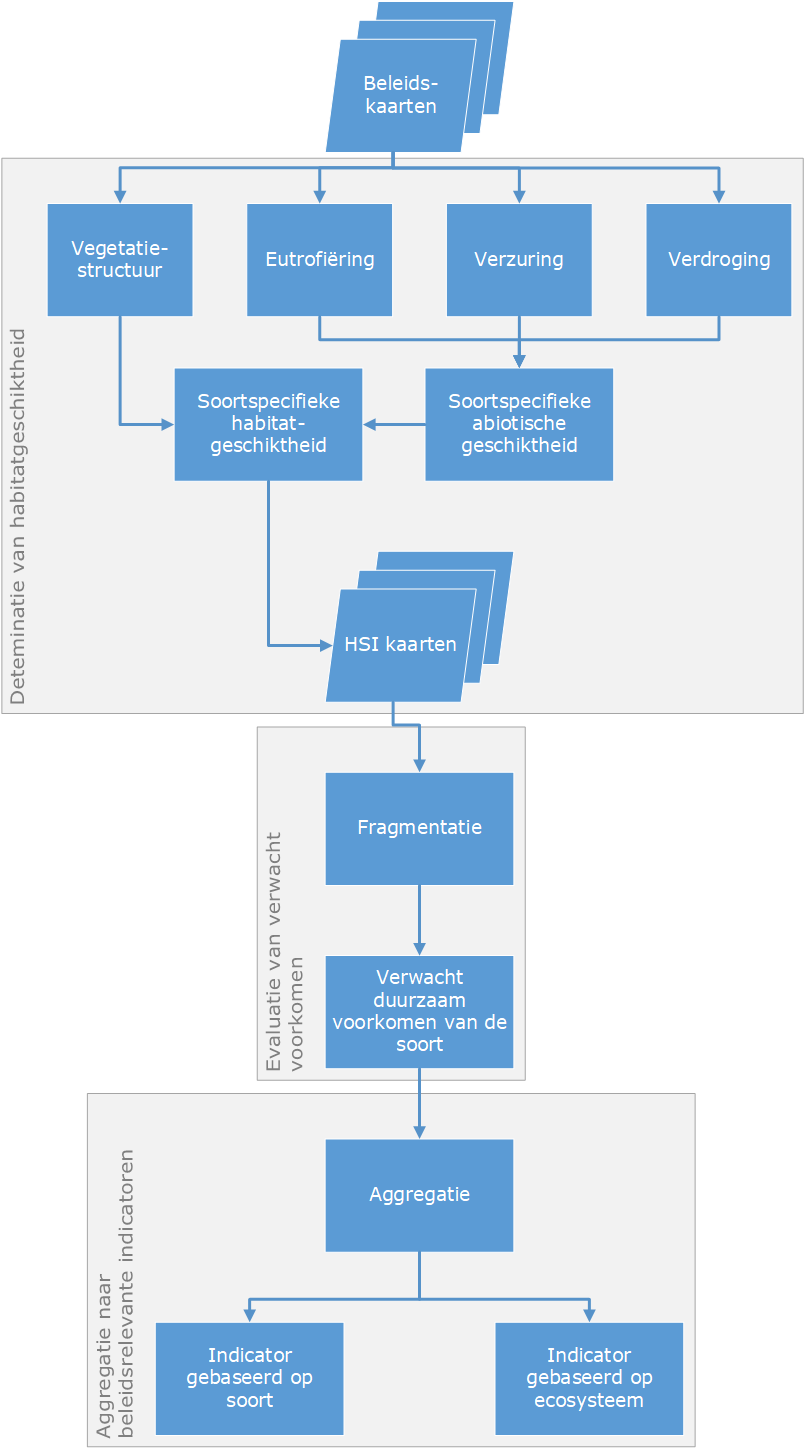
MNP berekent verwacht voorkomen eerst per soort (zie de species_models module) en aggregeert de resultaten van de individuele soorten daarna tot eindindicatoren (zie de subselection_evaluation en subselection_output modules.
Determineren van habitatgeschiktheid
MNP berekent de soortspecifieke habitatgeschiktheid volgens de volgende formule:
als
waarin \(\text{HSI}_i\) de Habitat Suitability Index van pixel \(i\) is, \(f(\text{VT}_x)\) de geschiktheid van vegetatietype/beheertype \(x\) bij optimale abiotische condities, \(\text{frac}_{x,i}\) de fractie vegatatietype/beheertype \(x\) in pixel \(i\) en \(\text{E}_i\) de geschiktheid van de abiotische condities van pixel \(i\). Wanneer een combinatie van \(f(\text{VT}_x)\text{E}_i\) lager is dan \(\text{HSI}_{min}\) wordt de combinatie als marginaal geschikt beschouwd en niet meegenomen in de totale \(\text{HSI}\) van die pixel. Voor de implementatie van de berekening, zie de API documentatie voor de habitat_suitability module.
De geschiktheid van de abiotische condities, ook wel de environmental factor \(\text{E}\), is een functie van de verschillende abiotische condities. De implementatie van MNP is flexibel in de formule voor \(\text{E}\). In de huidige versie van MNP wordt de volgende functie gebruikt:
waarbij \(f(\text{pH})\) de geschiktheid is op basis van de pH van de bodem, \(f(\text{Ndep})\) de geschiktheid op basis van de hoeveelheid stikstofdepositie en \(f(\text{GVG})\) de geschiktheid op basis van de gemiddelde voorjaarsgrondwaterstand. Hier gebruikt MNP dus een vermenigvuldiging van de individuele geschiktheden van de abiotische condities. Voor de implementatie van de berekening, zie de API documentatie voor de environmental_factor module.
Een alternatieve formule, op basis van het principe Liebig, waarbij de meest beperkende factor bepalend is, is ook bruikbaar. Verschillende vormen van deze methode zijn door Wamelink et al. (2022) getest maar bleken niet te leiden tot een verbetering van de soortmodellen.
De geschiktheid van de verschillende abiotische condities wordt gedetermineerd via trapsgewijze responscurven
(Pouwels et al., 2016):
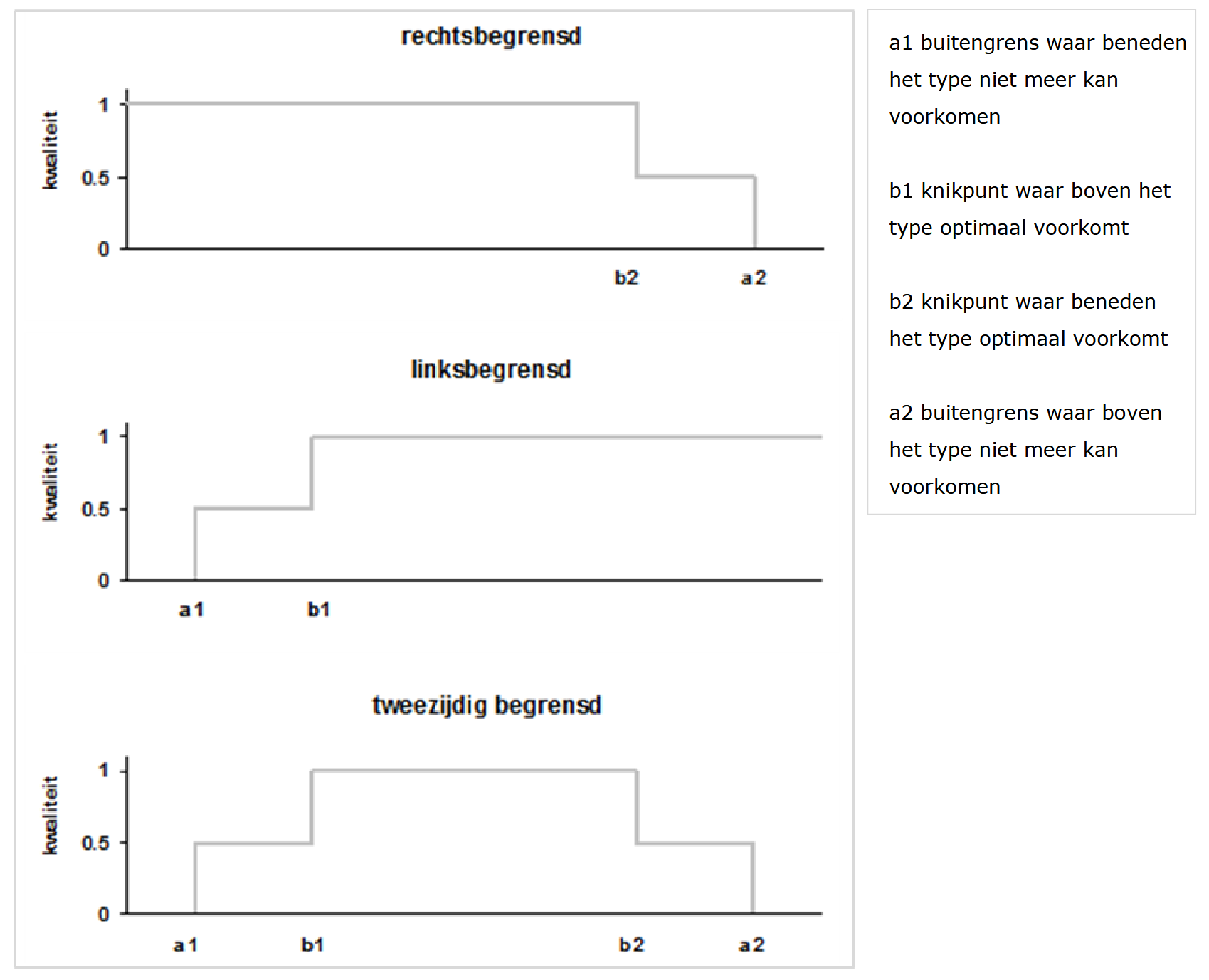
De HSI-kaarten voor de individuele soorten worden weggeschreven in de MNP cover directory.
Evaluatie van verwacht voorkomen
Wanneer voor een soort de habitatgeschiktheid is gedetermineerd, kan worden gekeken naar het effect van versnippering of fragmentatie van het geschikte habitat. Tot dusver was de analyse op basis van de lokale condities, nu wordt ook de ruimtelijke context meegenomen. Dit gebeurt in de volgende stappen:
HSI-kaart binair maken, waarbij 1 = habitat en 0 = geen habitat
Determineren welke stukken habitat binnen de lokale fusie-afstand of
local_distancevan elkaar vallen (lokale populaties), zie de API documentatie voor de clustering moduleDe voor geschiktheid gecorrigeerde som berekenen van het areaal habitat voor elke lokale populatie
Determineren welke lokale populaties groot genoeg zijn om een sleutelpopulatie of
key_populationte vormenDetermineren of er in Nederland voldoende sleutelpopulaties zijn om duurzaam te kunnen bestaan
Voor de implementatie van stappen 3, 4 en 5, zie de API documentatie voor de species_evaluation module.
Aggregatie naar beleidsrelevante indicatoren
Wanneer per soort is bepaald of de condities op orde zijn om duurzaam in Nederland voor te komen, kunnen de resultaten geaggregeerd worden.
De resultaten worden ongewogen geaggregeerd, elke soort weegt dus even zwaar mee.
Voor de soortenindicator wordt het aantal soorten dat duurzaam voor kan komen, gesommeerd. Deze som kan dan worden gedeeld door het aantal soorten dat is doorgerekend om tot het percentage doelbereik te komen.
Voor de ecosysteemindicator, ook wel fraction_key_populations, wordt per pixel gekeken voor hoeveel soorten deze pixel
onderdeel is van een sleutelpopulatie. Deze som wordt gedeeld door het aantal doorgerekende soorten dat voor die pixel
voor zou kunnen komen wanneer de drukfactoren (verzuring, eutrofiëring, verdroging en versnippering) niet mee zouden
worden genomen.
MNP is flexibel in welke soorten worden meegenomen in deze aggregatie. Standaard is dit de lijst met 146 soorten die typerend zijn voor de VHR én in de validatieronde van 2016 als goed genoeg gemodelleerd zijn gekwalificeerd. Ook kan worden gekozen om alleen een bepaalde soortgroep, bijvoorbeeld vlinders, of alleen stikstofgevoelige soorten te aggregeren. Bij elke MNP run kunnen meerdere soortselecties worden doorgerekend. Zie de API documentatie voor de subselection_evaluation en subselection_output modules voor meer detail.
Veranderingen tov. MNP 4.0
Extra functionaliteit
meerdere soortselecties doorrekenen in een run
optie om verschillende HSI procedures te definieren per soort
optie om verschillende land-type-experience procedures te definieren per soort
alle GIS output per soort wordt opgeleverd met een QGIS layer style file
gebruikers kunnen kiezen tussen default parameters en tabellen of eigen inbreng
Veranderingen onder de motorkap
De naamgeving van tabellen en kolommen is intuitiever gemaakt. Overzicht hiervan volgt later.
De vernieuwde versie van MNP gebruikt een nieuw clusteralgorithme, gebasseerd op dilation waarna vervolgens wordt gekeken of patches aan elkaar liggen. Hiervoor worden de morphology.disk en measure.label van skimage gebruikt.
Multicore aggregatie van landgebruikskaarten
Soortmodellen worden parallel doorgerekend
Vergelijking MNP 7.0 - MNP 4.0
Laten we kijken naar de verschillen tussen MNP in C++ de implementatie in python. Voor deze vergelijking zijn beide versies van MNP, uiteraard met dezelfde parameters en invoerkaarten, gedraaid voor de 146 soortenlijst.
HSI kaarten
Doordat het model veel verschillende berekeningen en vergelijkkingen doet is het onvermijdelijk dat er, als gevolg onderliggende techniek van de gebruikte software, minieme verschillen zitten in de resultaten van de berekeningen waarmee we tot de Habitat Suitability kaarten komen. We hebben dit zo veel mogelijk geprobeerd te beperken, het was niet mogelijk dit totaal weg te werken.
Omdat we de response van de soorten op omgevingsvariabelen met hele grove bell curves berekenen hebben deze verschillen in verreweg de meeste cellen geen consequenties. Echter, voor cellen waar de waarde erg dicht bij de knikpunten van de bell curves liggen kunnen wel verschillen optreden. Deze zijn dan ook meteen vrij groot als gevolg van de grofheid van de trappetjes’, maar al met al zijn de verschillen per kaart miniem, zie tabel hieronder. Voor het grootste deel van de soortmodellen is het verschil tussen MNP4.0 en de python-rebuild 0 cellen. Gezien er 145.600.000 cellen in een kaart zitten is dit zeer acceptabel. Het grootste verschil is met vijfhonderdste van een procent nog steeds verwaarloosbaar klein.
species code |
n cells different |
percentage of total cells |
|---|---|---|
S02000120 |
0 |
|
S02001040 |
2 |
0.0006 |
S02001730 |
0 |
|
S02001840 |
259 |
0.0150 |
S02002060 |
0 |
|
S02002310 |
15 |
0.0002 |
S02004560 |
0 |
|
S02004770 |
0 |
|
S02005190 |
415 |
0.0217 |
S02005410 |
0 |
|
S02005460 |
469 |
0.0174 |
S02007680 |
36 |
0.0092 |
S02008630 |
5 |
0.0001 |
S02008760 |
6 |
0.0001 |
S02009740 |
0 |
|
S02009760 |
383 |
0.0083 |
S02010050 |
0 |
|
S02010110 |
2 |
0.0001 |
S02011040 |
3 |
0.0002 |
S02011060 |
252 |
0.0149 |
S02011390 |
3 |
0.0001 |
S02011460 |
0 |
|
S02012360 |
87 |
0.0109 |
S02013080 |
6 |
0.0001 |
S02014420 |
4 |
0.0001 |
S02014790 |
6 |
0.0001 |
S02015080 |
3 |
0.0001 |
S02015200 |
0 |
|
S02017100 |
4 |
0.0001 |
S02017170 |
8 |
0.0001 |
S02018570 |
5 |
0.0001 |
S06000003 |
428 |
0.0200 |
S06000006 |
1 |
0.0001 |
S06000008 |
22 |
0.0209 |
S06000012 |
0 |
|
S06000035 |
60 |
0.0058 |
S06000042 |
0 |
|
S06000049 |
1 |
0.0001 |
S06000051 |
1 |
0.0002 |
S06000052 |
1 |
0.0000 |
S06000057 |
0 |
|
S06000064 |
61 |
0.0126 |
S06000065 |
0 |
|
S06000066 |
0 |
|
S06000076 |
0 |
|
S06000077 |
0 |
|
S06000080 |
0 |
|
S06000081 |
2 |
0.0001 |
S06000082 |
1 |
0.0001 |
S06000084 |
104 |
0.0125 |
S06000097 |
0 |
|
S06000099 |
0 |
|
S06000106 |
0 |
|
S06000107 |
0 |
|
S09000008 |
0 |
|
S09000034 |
0 |
|
S09000039 |
1 |
0.0004 |
S09000043 |
0 |
|
S09000053 |
0 |
|
S09000055 |
0 |
|
S09000076 |
0 |
|
S09000091 |
2 |
0.0010 |
S09000093 |
0 |
|
S09000100 |
0 |
|
S09000105 |
0 |
|
S09000148 |
57 |
0.0170 |
S09000157 |
0 |
|
S09000170 |
0 |
|
S09000196 |
24 |
0.0137 |
S09000217 |
0 |
|
S09000221 |
0 |
|
S09000230 |
66 |
0.0172 |
S09000231 |
4 |
0.0021 |
S09000236 |
7 |
0.0144 |
S09000255 |
0 |
|
S09000266 |
0 |
|
S09000271 |
48 |
0.0184 |
S09000284 |
0 |
|
S09000289 |
0 |
|
S09000324 |
0 |
|
S09000332 |
0 |
|
S09000341 |
0 |
|
S09000343 |
2 |
0.0010 |
S09000392 |
1 |
0.0016 |
S09000416 |
0 |
|
S09000418 |
0 |
|
S09000432 |
0 |
|
S09000438 |
0 |
|
S09000461 |
1 |
0.0003 |
S09000471 |
0 |
|
S09000472 |
0 |
|
S09000477 |
0 |
|
S09000479 |
6 |
0.0085 |
S09000481 |
0 |
|
S09000486 |
0 |
|
S09000491 |
0 |
|
S09000492 |
0 |
|
S09000496 |
227 |
0.0379 |
S09000497 |
0 |
|
S09000532 |
0 |
|
S09000558 |
1 |
0.0006 |
S09000560 |
0 |
|
S09000562 |
0 |
|
S09000566 |
17 |
0.0115 |
S09000568 |
21 |
0.0170 |
S09000595 |
8 |
0.0039 |
S09000597 |
0 |
|
S09000635 |
0 |
|
S09000668 |
0 |
|
S09000692 |
6 |
0.0063 |
S09000713 |
3 |
0.0038 |
S09000738 |
2 |
0.0016 |
S09000748 |
0 |
|
S09000754 |
0 |
|
S09000775 |
83 |
0.0241 |
S09000776 |
46 |
0.0095 |
S09000777 |
0 |
|
S09000827 |
0 |
|
S09000881 |
0 |
|
S09000891 |
0 |
|
S09000902 |
0 |
|
S09000913 |
0 |
|
S09000917 |
0 |
|
S09000924 |
0 |
|
S09000948 |
80 |
0.0481 |
S09000950 |
94 |
0.0383 |
S09000962 |
0 |
|
S09000963 |
41 |
0.0068 |
S09001011 |
0 |
|
S09001034 |
40 |
0.0131 |
S09001038 |
1 |
0.0010 |
S09001110 |
3 |
0.0014 |
S09001111 |
47 |
0.0148 |
S09001128 |
0 |
|
S09001138 |
0 |
|
S09001150 |
0 |
|
S09001158 |
2 |
0.0083 |
S09001181 |
10 |
0.0052 |
S09001205 |
9 |
0.0254 |
S09001258 |
3 |
0.0005 |
S09001283 |
17 |
0.0114 |
S09001312 |
0 |
|
S09001332 |
113 |
0.0307 |
S09001364 |
0 |
|
S09001380 |
0 |
|
S09001953 |
0 |
Tabel 1 Het verschil tussen MNP60 en rebuild in aantal cellen in aantal cellen en als fractie van het aantal bezette cellen is. Als we zouden kijken naar het aantal cellen dat anders is als fractie van het totaal aantal cellen in een kaart zouden alle verschillen 0 procent zijn.
Output tabellen en het percentage
Het grootste verschil in output tussen beide modelversies komt door het clusteralgorithme. MNP 4.0 gebruikte daarvoor het LARCH model. De python versie van MNP gebruikt een nieuw clusteralgorithme, gebasseerd op dilation waarna wordt gekeken of patches aan elkaar liggen. Hiervoor worden de morphology.disk en measure.label van skimage gebruikt.
LARCH clustert minder patches aan elkaar waardoor het aantal populaties voor een soortmodel in MNP60 hoger is dan in rebuild. In onderstaande tabel staat hoe de output tabellen van het oude en nieuwe model van elkaar verschillen.
scientific name |
N populations |
effective area(m2) |
keypopulations |
normkeys |
|---|---|---|---|---|
Apium graveolens |
0.1910 |
0.0000 |
0.0000 |
0.0000 |
Carex extensa |
0.1883 |
0.0000 |
0.0000 |
-0.0013 |
Blysmus rufus |
0.1778 |
0.0000 |
0.0000 |
0.0000 |
Atriplex pedunculata |
0.1360 |
0.0000 |
0.0000 |
0.0000 |
Liparis loeselii |
0.1261 |
0.0002 |
0.0002 |
-0.0588 |
Limonium vulgare |
0.1246 |
0.0000 |
0.0000 |
0.0000 |
Cochlearia officinalis subsp. anglica |
0.1164 |
0.0000 |
0.0000 |
-0.0013 |
Schoenus nigricans |
0.1148 |
0.0027 |
0.0027 |
0.0013 |
Emberiza citrinella |
0.1041 |
-0.0004 |
-0.0004 |
-0.0106 |
Eleocharis quinqueflora |
0.1034 |
-0.0059 |
-0.0059 |
0 |
Lycopodiella inundata |
0.1016 |
-0.0103 |
-0.0103 |
-0.0500 |
Drosera longifolia |
0.0943 |
-0.0133 |
-0.0133 |
0.0000 |
Pyrola rotundifolia |
0.0923 |
-0.0003 |
-0.0003 |
0.0000 |
Radiola linoides |
0.0907 |
-0.0183 |
-0.0183 |
-0.0250 |
Hammarbya paludosa |
0.0865 |
-0.0040 |
-0.0040 |
0.0000 |
Epipactis palustris |
0.0855 |
-0.0034 |
-0.0034 |
-0.0234 |
Lullula arborea |
0.0844 |
0.0000 |
0.0000 |
0.5385 |
Equisetum variegatum |
0.0840 |
-0.0076 |
-0.0076 |
0 |
Drosera rotundifolia |
0.0821 |
-0.0065 |
-0.0065 |
-0.0083 |
Sitta europaea |
0.0817 |
-0.0002 |
-0.0002 |
-0.0040 |
Parus montanus |
0.0810 |
-0.0010 |
-0.0010 |
0.3333 |
Dendrocopos major |
0.0769 |
-0.0001 |
-0.0001 |
-0.0089 |
Sagina nodosa |
0.0758 |
0.0000 |
0.0000 |
0.0000 |
Pyrrhula pyrrhula ssp. europoea |
0.0747 |
-0.0046 |
-0.0046 |
-0.0226 |
Dryocopus martius |
0.0720 |
0.0001 |
0.0001 |
0.0885 |
Phylloscopus sibilatrix |
0.0711 |
-0.0006 |
-0.0006 |
-0.0056 |
Gentiana pneumonanthe |
0.0695 |
-0.0088 |
-0.0088 |
-0.0200 |
Vaccinium oxycoccos |
0.0686 |
-0.0195 |
-0.0195 |
-0.0313 |
Coccothraustes coccothraustes |
0.0681 |
-0.0041 |
-0.0041 |
0.0086 |
Eriophorum vaginatum |
0.0674 |
-0.0193 |
-0.0193 |
-0.0141 |
Orobanche picridis |
0.0653 |
0.0001 |
0.0001 |
0 |
Andromeda polifolia |
0.0634 |
-0.0222 |
-0.0222 |
0.0000 |
Erodium lebelii |
0.0623 |
-0.0044 |
-0.0044 |
-0.0029 |
Platanthera bifolia |
0.0618 |
-0.0072 |
-0.0072 |
-0.0172 |
Lycaena dispar |
0.0617 |
0.0000 |
0.0000 |
-0.2500 |
Cirsium dissectum |
0.0613 |
-0.0110 |
-0.0110 |
0.0000 |
Eryngium maritimum |
0.0604 |
0.0000 |
0.0000 |
0.0000 |
Carex pulicaris |
0.0596 |
-0.0074 |
-0.0074 |
-0.0071 |
Bromopsis erecta |
0.0593 |
-0.0006 |
-0.0006 |
0 |
Valeriana dioica |
0.0584 |
-0.0068 |
-0.0068 |
0.0000 |
Fritillaria meleagris |
0.0551 |
-0.0005 |
-0.0005 |
0.0000 |
Thymus pulegioides |
0.0546 |
-0.0143 |
-0.0143 |
-0.0163 |
Carex hostiana |
0.0532 |
-0.0097 |
-0.0097 |
-0.0400 |
Silene otites |
0.0524 |
-0.0002 |
-0.0002 |
0.0000 |
Milium vernale |
0.0519 |
-0.0059 |
-0.0059 |
0 |
Succisa pratensis |
0.0501 |
-0.0096 |
-0.0096 |
-0.0145 |
Trisetum flavescens |
0.0490 |
-0.0052 |
-0.0052 |
-0.0064 |
Lycopodium tristachyum |
0.0481 |
0.0016 |
0.0016 |
0.0000 |
Carex diandra |
0.0480 |
-0.0047 |
-0.0047 |
0 |
Sedum sexangulare |
0.0473 |
-0.0071 |
-0.0071 |
-0.0073 |
Anthus pratensis |
0.0469 |
-0.0003 |
-0.0003 |
-0.0571 |
Botrychium lunaria |
0.0451 |
-0.0007 |
-0.0007 |
-0.0004 |
Centaurea scabiosa |
0.0449 |
-0.0038 |
-0.0038 |
0.0000 |
Lycopodium clavatum |
0.0438 |
-0.0051 |
-0.0051 |
-0.0050 |
Oriolus oriolus |
0.0426 |
-0.0092 |
-0.0092 |
0.0000 |
Seriphidium maritimum |
0.0425 |
0.0000 |
0.0000 |
-0.0003 |
Ophrys insectifera |
0.0414 |
-0.0024 |
-0.0024 |
0.0000 |
Euphorbia palustris |
0.0413 |
-0.0004 |
-0.0004 |
-0.0009 |
Callophrys rubi |
0.0413 |
0.0000 |
0.0000 |
-0.0076 |
Carum carvi |
0.0408 |
-0.0035 |
-0.0035 |
-0.0078 |
Alauda arvensis |
0.0405 |
-0.0014 |
-0.0014 |
-0.0351 |
Campanula rapunculus |
0.0398 |
-0.0064 |
-0.0064 |
0.0000 |
Sanicula europaea |
0.0383 |
-0.0055 |
-0.0055 |
-0.0017 |
Hipparchia semele |
0.0383 |
-0.0048 |
-0.0048 |
-0.0032 |
Euphorbia paralias |
0.0377 |
0.0000 |
0.0000 |
0.0000 |
Pernis apivorus |
0.0376 |
0.0001 |
0.0001 |
0.0500 |
Carex ericetorum |
0.0376 |
-0.0009 |
-0.0009 |
0.0000 |
Armeria maritima |
0.0364 |
0.0000 |
0.0000 |
0.0000 |
Salvia pratensis |
0.0363 |
-0.0042 |
-0.0042 |
-0.0077 |
Daphne mezereum |
0.0354 |
-0.0051 |
-0.0051 |
0 |
Potentilla sterilis |
0.0352 |
-0.0113 |
-0.0113 |
0 |
Asparagus officinalis subsp. prostratus |
0.0347 |
0.0000 |
0.0000 |
0 |
Plantago maritima |
0.0340 |
0.0002 |
0.0002 |
0.0002 |
Actaea spicata |
0.0331 |
0.0000 |
0.0000 |
0.0000 |
Knautia arvensis |
0.0319 |
-0.0044 |
-0.0044 |
-0.0020 |
Eriophorum gracile |
0.0309 |
-0.0028 |
-0.0028 |
0 |
Veronica austriaca subsp. teucrium |
0.0298 |
-0.0042 |
-0.0042 |
-0.0263 |
Orchis purpurea |
0.0296 |
-0.0008 |
-0.0008 |
0.0385 |
Sagina maritima |
0.0275 |
-0.0009 |
-0.0009 |
-0.0016 |
Alopecurus bulbosus |
0.0269 |
0.0000 |
0.0000 |
0.0000 |
Cochlearia officinalis subsp. officinalis |
0.0263 |
0.0000 |
0.0000 |
0.0002 |
Lanius excubitor |
0.0250 |
0.0000 |
0.0000 |
0 |
Hordeum marinum |
0.0240 |
0.0000 |
0.0000 |
0.0000 |
Cephalanthera damasonium |
0.0232 |
0.0000 |
0.0000 |
0 |
Althaea officinalis |
0.0223 |
0.0000 |
0.0000 |
0.5714 |
Bupleurum tenuissimum |
0.0213 |
0.0000 |
0.0000 |
0.0000 |
Anthus campestris |
0.0189 |
-0.0002 |
-0.0002 |
0 |
Recurvirostra avosetta |
0.0170 |
0.0000 |
0.0000 |
0.0000 |
Tringa totanus |
0.0167 |
0.0001 |
0.0001 |
-0.0161 |
Charadrius alexandrinus |
0.0161 |
0.0000 |
0.0000 |
0.0000 |
Gallinago gallinago |
0.0130 |
0.0002 |
0.0002 |
0.9000 |
Numenius arquata |
0.0129 |
0.0000 |
0.0000 |
0.9216 |
Asio flammeus |
0.0090 |
0.0000 |
0.0000 |
0 |
Anas crecca |
0.0079 |
-0.0010 |
-0.0010 |
0.0000 |
Tadorna tadorna |
0.0075 |
-0.0001 |
-0.0001 |
0.0000 |
Podiceps nigricollis |
0.0065 |
-0.0022 |
-0.0022 |
0.0000 |
Nycticorax nycticorax |
0.0063 |
-0.0014 |
-0.0014 |
0.0000 |
Genista pilosa |
0.0015 |
-0.0065 |
-0.0065 |
-0.0066 |
Saxicola rubicola |
0.0001 |
-0.0049 |
-0.0049 |
4.6667 |
Allium ursinum |
0.0001 |
-0.0038 |
-0.0038 |
0 |
Somateria mollissima |
0.0000 |
-0.0001 |
-0.0001 |
0.0000 |
Luscinia megarhynchos |
0.0000 |
-0.0010 |
-0.0010 |
0.0000 |
Luscinia svecica |
0.0000 |
0.0001 |
0.0001 |
0.0000 |
Oenanthe oenanthe |
0.0000 |
-0.0033 |
-0.0033 |
2.0000 |
Locustella naevia |
0.0000 |
0.0001 |
0.0001 |
0.0000 |
Thymelicus sylvestris |
0.0000 |
-0.0011 |
-0.0011 |
-0.0016 |
Hesperia comma |
0.0000 |
-0.0010 |
-0.0010 |
0.0000 |
Erynnis tages |
0.0000 |
0.0002 |
0.0002 |
0.0000 |
Pyrgus malvae |
0.0000 |
-0.0025 |
-0.0025 |
0.0000 |
Plebeius argus |
0.0000 |
-0.0041 |
-0.0041 |
-0.0078 |
Plebeius optilete |
0.0000 |
-0.0003 |
-0.0003 |
0.0000 |
Aricia agestis |
0.0000 |
0.0000 |
0.0000 |
0.0000 |
Maculinea alcon |
0.0000 |
-0.0039 |
-0.0039 |
0.0000 |
Limenitis camilla |
0.0000 |
0.0001 |
0.0001 |
0.0000 |
Limenitis populi |
0.0000 |
0.0000 |
0.0000 |
0.0000 |
Apatura iris |
0.0000 |
0.0000 |
0.0000 |
0.0000 |
Boloria aquilonaris |
0.0000 |
-0.0084 |
-0.0084 |
0.0000 |
Boloria selene |
0.0000 |
-0.0006 |
-0.0006 |
0.0000 |
Brenthis ino |
0.0000 |
0.0000 |
0.0000 |
0.0000 |
Issoria lathonia |
0.0000 |
-0.0012 |
-0.0012 |
-0.0012 |
Argynnis niobe |
0.0000 |
-0.0129 |
-0.0129 |
0.0000 |
Argynnis aglaja |
0.0000 |
-0.0087 |
-0.0087 |
0.0000 |
Coenonympha arcania |
0.0000 |
-0.0127 |
-0.0127 |
0.0000 |
Coenonympha tullia ssp. tullia |
0.0000 |
-0.0060 |
-0.0060 |
0.0000 |
Hipparchia statilinus |
0.0000 |
-0.0166 |
-0.0166 |
0 |
Anagallis tenella |
0.0000 |
-0.0086 |
-0.0086 |
0 |
Arnica montana |
0.0000 |
-0.0049 |
-0.0049 |
0.0000 |
Carex buxbaumii |
0.0000 |
-0.0098 |
-0.0098 |
0 |
Carex trinervis |
0.0000 |
-0.0041 |
-0.0041 |
0.0000 |
Cicendia filiformis |
0.0000 |
-0.0051 |
-0.0051 |
0.0000 |
Elatine hexandra |
0.0000 |
-0.0070 |
-0.0070 |
0 |
Erica cinerea |
0.0000 |
-0.0009 |
-0.0009 |
0.0000 |
Euphorbia amygdaloides |
0.0000 |
0.0000 |
0.0000 |
0 |
Euphorbia cyparissias |
0.0000 |
-0.0129 |
-0.0129 |
-0.0174 |
Genista anglica |
0.0000 |
-0.0020 |
-0.0020 |
-0.0006 |
Gentiana cruciata |
0.0000 |
-0.0005 |
-0.0005 |
0.0000 |
Isoetes echinospora |
0.0000 |
0.0000 |
0.0000 |
0 |
Lathyrus nissolia |
0.0000 |
0.0000 |
0.0000 |
0 |
Lobelia dortmanna |
0.0000 |
-0.0204 |
-0.0204 |
0 |
Parapholis strigosa |
0.0000 |
0.0000 |
0.0000 |
0.0000 |
Pedicularis sylvatica |
0.0000 |
-0.0044 |
-0.0044 |
-0.0030 |
Polygala serpyllifolia |
0.0000 |
-0.0030 |
-0.0030 |
0.0000 |
Polygala vulgaris |
0.0000 |
-0.0016 |
-0.0016 |
-0.0017 |
Viola canina |
0.0000 |
-0.0032 |
-0.0032 |
-0.0037 |
Thalictrum minus |
0.0000 |
-0.0027 |
-0.0027 |
0.0000 |
Gentianella amarella |
-0.0004 |
-0.0001 |
-0.0001 |
0 |
Tabel 2 Verschil in waarden van soortmodellen in rebuild en opzichte van MNP60. Positieve waarden betekenen een toename in rebuild ten opzichte van MNP60 en negatieve waarden een afname. De velduil (Asio flammeus_) laat een verschil in normkeys zien van -Infinity, dit komt doordat er in rebuild 1 sleutelpopulatie wordt geclusterd versus 0 in MNP60, dit geeft dus een fractionele toename van oneindig.
De verschillen in totale oppervlakte, en dus ook sleutelpopulaties, per soort zijn dus heel erg klein. Dat is mooi, maar dit zijn getallen die berekend worden over de hele kaart, onafhankelijk van clustering. Uit de tabel hieronder blijkt dat slechts twee soorten van duurzaamheidsklasse veranderen! Als we naar deze soorten kijken zien we niet dat ze een uitzonderlijk groot verschil in effectief oppervlakte, maar wel in normkeys.
scientific name |
duurzaamheidsklasse MNP 4.0 |
Duurzaamheidsklasse MNP v7.0 |
|---|---|---|
Lullula arborea |
2 |
3 |
Saxicola rubicola |
1 |
2 |
Omdat slechts twee soorten van duurzaamheidsklasse zijn gewisseld, waarvan er maar ��n van klasse twee naar klasse drie is gegaan, is het verschil in de duurzaamheidspercentages klein: 57.53% in MNP4.0 vs 58.22% in MNP v7.0.
Overzicht van vogels, planten en vlinders in MNP
Op deze pagina bespreken we de plant-, vogel- en vlindersoorten die gemodelleerd worden door MNP. Om MNP te gebruiken, geeft de gebruiker een lijst met soorten op waarvoor hij/zij de uitkomsten wil zien. MNP controleert elke gevraagde soort op twee aspecten:
is het uberhaupt een soort?
zijn de benodigde parameters bekend voor deze soort?
Voor de eerste controle refereert MNP naar de species names tabel. Deze tabel benoemt 812 planten, 56 vlinders en 150 vogels: 1018 soorten in totaal. Deze lijst is opgesteld eind 2015 en bevat: ‘SNL kwaliteitssoorten en NDT doelsoorten’ (Marjolein van Adrichem). In 2021 zijn daar 60 stikstof-gevoellige plantensoorten toegevoegd (110 plantsoorten die reeds in de lijst stonden zijn eveneens als N-gevoellig aangemerkt).
Voor de tweede controle refereert MNP aan de parametertabellen, zoals de GVG respons tabel en de species traits tabel. Het MNP team heeft deze tabellen in de loop van de jaren opgesteld en ontwikkeld. De tabellen bevatten niet alle soorten uit de eerder genoemde ‘species names’ tabel. Bijvoorbeeld, NDep-respons is bekend voor slechts 967 soorten (116 vogels, 51 vlinders en 809 planten).
De valid species zijn dan de soorten die in alle tabellen genoemd zijn en waar MNP in principe mee kan rekenen. In het Evaluatie Natuurpact 2016 project is gerekend met deze 468 soorten. Handmatig is beoordeeld of MNP deze soorten ‘goed’ modeleerde. Dit was het geval voor 281 soorten. Van deze 281 zijn er 146 aangemerkt als ‘typerende soorten voor beschermde habitats uit de Habitatrichtlijn’ (Pouwel etal 2017, pp 41).
De 146 soortenlijst wordt het vaakst gebruikt in MNP analyses. Het gaat om 31 vogels, 23 vlinders en 92 planten:
scientific name |
local name |
|---|---|
Actaea spicata |
Christoffelkruid |
Allium ursinum |
Daslook |
Alopecurus bulbosus |
Knolvossenstaart |
Althaea officinalis |
Heemst |
Anagallis tenella |
Teer guichelheil |
Andromeda polifolia |
Lavendelhei |
Apium graveolens |
Selderij |
Armeria maritima |
Engels gras |
Arnica montana |
Valkruid |
Seriphidium maritimum |
Zeealsem |
Asparagus officinalis subsp. prostratus |
Liggende asperge |
Atriplex pedunculata |
Gesteelde zoutmelde |
Blysmus rufus |
Rode bies |
Botrychium lunaria |
Gelobde maanvaren |
Bromopsis erecta |
Bergdravik |
Bupleurum tenuissimum |
Fijn goudscherm |
Campanula rapunculus |
Rapunzelklokje |
Carex buxbaumii |
Knotszegge |
Carex diandra |
Ronde zegge |
Carex ericetorum |
Heidezegge |
Carex extensa |
Kwelderzegge |
Carex hostiana |
Blonde zegge |
Carex pulicaris |
Vlozegge |
Carex trinervis |
Drienervige zegge |
Carum carvi |
Karwij |
Centaurea scabiosa |
Grote centaurie |
Cephalanthera damasonium |
Bleek bosvogeltje |
Cicendia filiformis |
Draadgentiaan |
Cirsium dissectum |
Spaanse ruiter |
Cochlearia officinalis subsp. anglica |
Engels lepelblad |
Cochlearia officinalis subsp. officinalis |
Echt lepelblad |
Daphne mezereum |
Rood peperboompje |
Drosera longifolia |
Lange zonnedauw |
Drosera rotundifolia |
Ronde zonnedauw |
Elatine hexandra |
Gesteeld glaskroos |
Eleocharis quinqueflora |
Armbloemige waterbies |
Epipactis palustris |
Moeraswespenorchis |
Equisetum variegatum |
Bonte paardenstaart |
Erica cinerea |
Rode dophei |
Eriophorum gracile |
Slank wollegras |
Eriophorum vaginatum |
Eenarig wollegras |
Erodium lebelii |
Kleverige reigersbek |
Eryngium maritimum |
Blauwe zeedistel |
Euphorbia amygdaloides |
Amandelwolfsmelk |
Euphorbia cyparissias |
Cipreswolfsmelk |
Euphorbia palustris |
Moeraswolfsmelk |
Euphorbia paralias |
Zeewolfsmelk |
Fritillaria meleagris |
Wilde kievitsbloem |
Genista anglica |
Stekelbrem |
Genista pilosa |
Kruipbrem |
Gentiana cruciata |
Kruisbladgentiaan |
Gentiana pneumonanthe |
Klokjesgentiaan |
Gentianella amarella |
Slanke gentiaan |
Hammarbya paludosa |
Veenmosorchis |
Hordeum marinum |
Zeegerst |
Isoetes echinospora |
Kleine biesvaren |
Knautia arvensis |
Beemdkroon |
Lathyrus nissolia |
Graslathyrus |
Limonium vulgare |
Lamsoor |
Liparis loeselii |
Groenknolorchis |
Lobelia dortmanna |
Waterlobelia |
Lycopodiella inundata |
Moeraswolfsklauw |
Lycopodium clavatum |
Grote wolfsklauw |
Lycopodium tristachyum |
Kleine wolfsklauw |
Milium vernale |
Ruw gierstgras |
Ophrys insectifera |
Vliegenorchis |
Orchis purpurea |
Purperorchis |
Orobanche picridis |
Bitterkruidbremraap |
Parapholis strigosa |
Dunstaart |
Pedicularis sylvatica |
Heidekartelblad |
Plantago maritima |
Zeeweegbree |
Platanthera bifolia |
Welriekende nachtorchis |
Polygala serpyllifolia |
Liggende vleugeltjesbloem |
Polygala vulgaris |
Gewone vleugeltjesbloem |
Potentilla sterilis |
Aardbeiganzerik |
Pyrola rotundifolia |
Rond wintergroen |
Radiola linoides |
Dwergvlas |
Sagina maritima |
Zeevetmuur |
Sagina nodosa |
Sierlijke vetmuur |
Salvia pratensis |
Veldsalie |
Sanicula europaea |
Heelkruid |
Schoenus nigricans |
Knopbies |
Sedum sexangulare |
Zacht vetkruid |
Silene otites |
Oorsilene |
Succisa pratensis |
Blauwe knoop |
Thalictrum minus |
Kleine ruit |
Thymus pulegioides |
Grote tijm |
Trisetum flavescens |
Goudhaver |
Vaccinium oxycoccos |
Kleine veenbes |
Valeriana dioica |
Kleine valeriaan |
Veronica austriaca subsp. teucrium |
Brede ereprijs |
Viola canina |
Hondsviooltje |
Apatura iris |
Grote weerschijnvlinder |
Argynnis aglaja |
Grote parelmoervlinder |
Argynnis niobe |
Duinparelmoervlinder |
Aricia agestis |
Bruin blauwtje |
Boloria aquilonaris |
Veenbesparelmoervlinder |
Boloria selene |
Zilveren maan |
Brenthis ino |
Purperstreepparelmoervlinder |
Callophrys rubi |
Groentje |
Coenonympha arcania |
Tweekleurig hooibeestje |
Coenonympha tullia ssp. tullia |
Veenhooibeestje |
Erynnis tages |
Bruin dikkopje |
Hesperia comma |
Kommavlinder |
Hipparchia semele |
Heivlinder |
Hipparchia statilinus |
Kleine heivlinder |
Issoria lathonia |
Kleine parelmoervlinder |
Limenitis camilla |
Kleine ijsvogelvlinder |
Limenitis populi |
Grote ijsvogelvlinder |
Lycaena dispar |
Grote vuurvlinder |
Maculinea alcon |
Gentiaanblauwtje |
Plebeius argus |
Heideblauwtje |
Plebeius optilete |
Veenbesblauwtje |
Pyrgus malvae |
Aardbeivlinder |
Thymelicus sylvestris |
Geelsprietdikkopje |
Alauda arvensis |
Veldleeuwerik |
Anas crecca |
Wintertaling |
Anthus campestris |
Duinpieper |
Anthus pratensis |
Graspieper |
Asio flammeus |
Velduil |
Charadrius alexandrinus |
Strandplevier |
Coccothraustes coccothraustes |
Appelvink |
Dendrocopos major |
Grote bonte specht |
Dryocopus martius |
Zwarte specht |
Emberiza citrinella |
Geelgors |
Gallinago gallinago |
Watersnip |
Lanius excubitor |
Klapekster |
Locustella naevia |
Sprinkhaanzanger |
Lullula arborea |
Boomleeuwerik |
Luscinia megarhynchos |
Nachtegaal |
Luscinia svecica |
Blauwborst |
Numenius arquata |
Wulp |
Nycticorax nycticorax |
Kwak |
Oenanthe oenanthe |
Tapuit |
Oriolus oriolus |
Wielewaal |
Parus montanus |
Matkop |
Pernis apivorus |
Wespendief |
Phylloscopus sibilatrix |
Fluiter |
Podiceps nigricollis |
Geoorde fuut |
Pyrrhula pyrrhula ssp. europoea |
Midden-Europese goudvink |
Recurvirostra avosetta |
Kluut |
Saxicola rubicola |
Roodborsttapuit |
Sitta europaea |
Boomklever |
Somateria mollissima |
Eider |
Tadorna tadorna |
Bergeend |
Tringa totanus |
Tureluur |